Il DNA cos’è? Si tratta di una molecola che contiene tutte le informazioni relative alla sintesi delle proteine. Per questo motivo viene spesso definita come il “libro delle istruzioni” del nostro organismo, come di tutti gli altri sistemi viventi. Ma com’è fatta questa molecola così speciale?
IN BREVE
IL DNA COS’È?
Il DNA cos’è? Il DNA, o acido desossiribonucleico, è un polimero organico i cui monomeri sono i nucleotidi. Ogni nucleotide è costituito da un gruppo fosfato, una base azotata ed una molecola di desossiribosio, la quale è formata da uno zucchero pentoso ciclico privato di un atomo di ossigeno. Le basi azotate presenti nel DNA sono 4, vale a dire citosina (C), guanina (G), timina (T) e adenina (A).
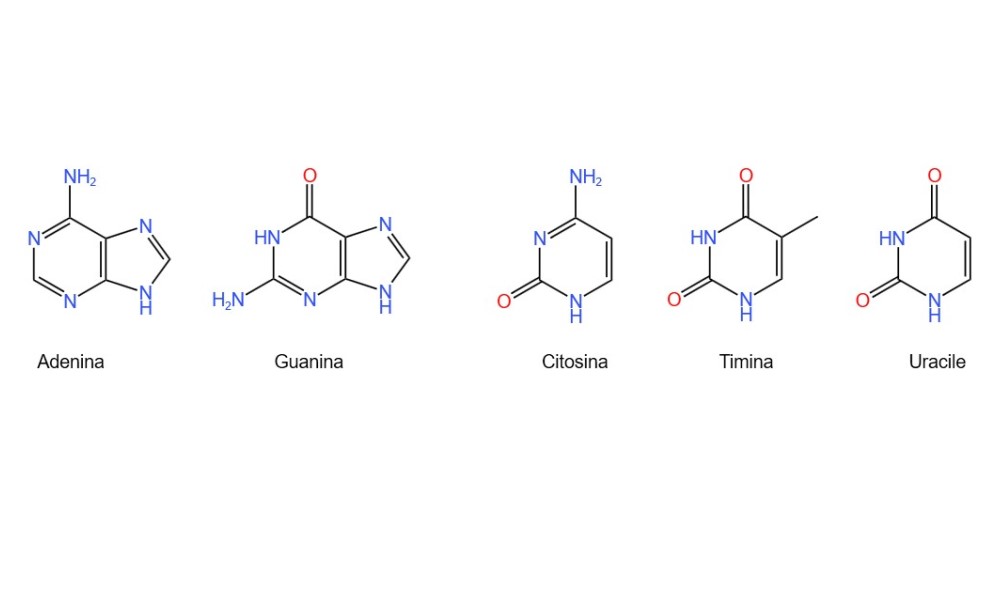
L’informazione necessaria per la sintesi proteica è codificata nella sequenza dei nucleotidi che formano il DNA. L’insieme delle regole con le quali questa viene tradotta costituisce quello che viene chiamato il codice genetico.
Come già detto, ogni monomero è costituito da una base azotata, un gruppo fosfato ed una molecola di 2-deossiribosio. Il gruppo fosfato è legato al carbonio dello zucchero in posizione 5′, mentre le basi azotate sono legate al carbonio in posizione 1’ attraverso un legame N-β-glicosidico. Questo legame si forma tra un atomo di azoto della base azotata e il carbonio 1’ dello zucchero.
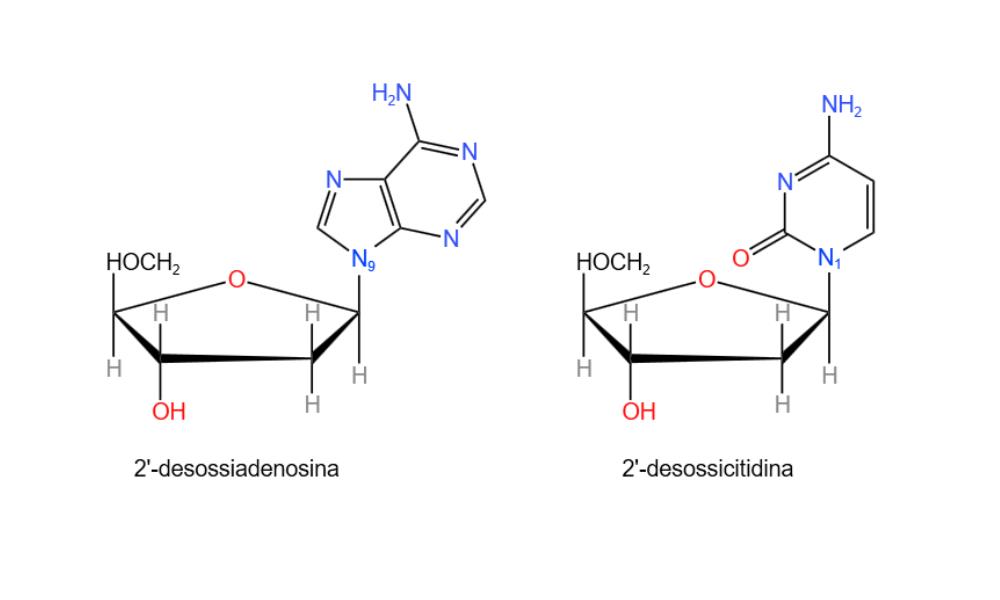
La struttura primaria del DNA è determinata quindi dalla sequenza dei nucleotidi, i quali sono legati tra loro attraverso i gruppi fosfato. Ogni gruppo fosfato è legato a due molecole di desossiribosio: ad una è legato in posizione 5’, mentre all’altra è legato in posizione 3’.
LA STRUTTURA DEL DNA
Sin dal 1938 è noto, grazie alla tecnica della diffrazione ai raggi X, che il DNA ha una struttura molecolare ben precisa. Nel 1950 Erwin Chargaff, analizzando il DNA di animali diversi, scoprì che il rapporto tra la quantità di adenina e timina ed il rapporto tra citosina e guanina era sempre uguale a 1. Nel 1953 Watson e Crick (che successivamente vinsero il Nobel per la medicina) riuscirono, grazie alle scoperte precedenti, a formulare una teoria plausibile per la struttura del DNA.
Nella teoria di Watson e Crick il DNA presenta una struttura a doppia elica. Il DNA è quindi costituito da due catene polinucleotidiche antiparallele avvolte intorno ad un asse comune, che formano quindi le due eliche destrorse. Le due eliche sono tenute insieme dai legami a idrogeno che si vengono a formare tra le basi azotate giacenti perpendicolari al piano dell’elica. Tuttavia, è presente una complementarietà tra le basi: l’adenina forma ponti idrogeno solo con la timina, e la citosina solo con la guanina. Il modello a doppia elica prevede un diametro pari a 20 Å, che le coppie di basi azotate adiacenti siano distanti 3,4 Å e che le coppie si ripetano ogni rotazione di 36°. Questo implica che per ogni giro completo dell’elica ci siano 10 coppie di basi azotate.
Recentemente è emerso che la struttura proposta da Watson e Crick è soltanto una tra le possibili strutture del DNA. Attualmente le diverse strutture del DNA vengono classificate in tre diverse categorie: A, B e Z. La struttura scoperta da Watson e Crick non è altro che la forma B, ovvero la più comune.
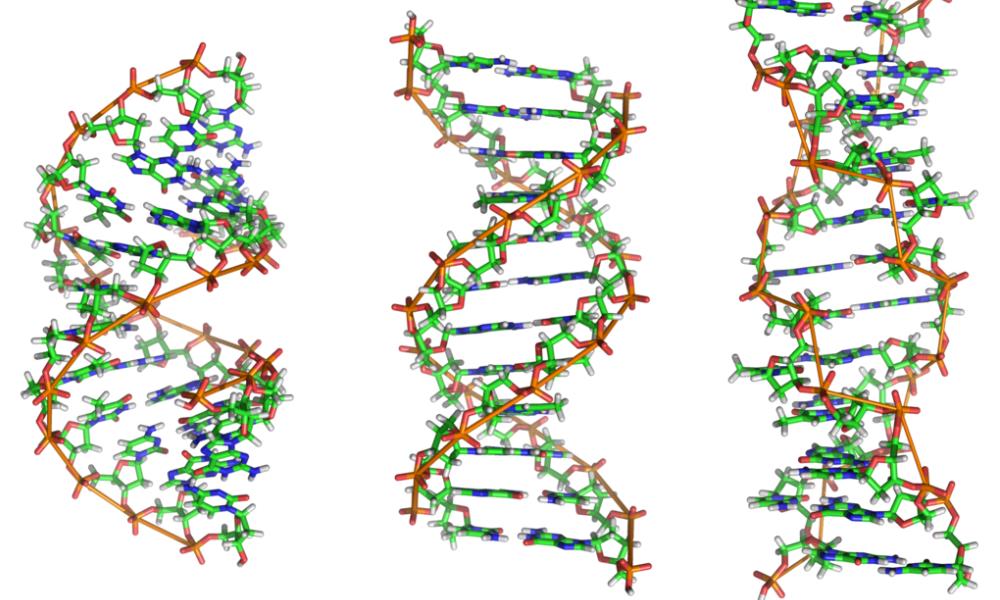
COSA FA IL DNA?
Con il modello a doppia elica proposto da Watson e Creek è stato possibile comprendere come avviene la replicazione del DNA. Durante questo processo avviene la rottura dei legami idrogeno tra le basi azotate, cosa che provoca la separazione dei due filamenti polinucleotidici. A questo punto i due filamenti srotolati sono in grado di legare nuovi nucleotidi. Questi, però, giungono nel nucleo sottoforma di nucleosidi trifosfati. Ciò significa che questi nucleotidi presentano tre gruppi fosfato consecutivi anziché uno solo. Nella replicazione del DNA intervengono diversi enzimi: per esempio, la DNA polimerasi si occupa di legare i nucleotidi ad uno dei due filamenti stampo. La DNA ligasi, invece, permette la formazione dei legami tra i nucleotidi aggiunti dalla DNA polimerasi.
Affinché avvenga la sintesi proteica è necessario che avvenga la trascrizione del DNA in RNA messaggero. Questo passaggio è fondamentale per la sintesi delle proteine perché è l’mRNA a interagire con i ribosomi, e non il DNA. Una serie di tre basi azotate sull’RNA codifica un amminoacido, e la sequenza di amminoacidi costituisce poi la proteina. Si dice che il codice genetico è degenere, poiché sono disponibili 20 amminoacidi distinti, per 64 possibili combinazioni di basi azotate.
Per comprendere meglio le informazioni contenute nel DNA è necessario effettuare un’operazione di sequenziamento. L’operazione è tutt’altro che semplice, dato che anche le più piccole molecole di DNA contengono almeno 5000 nucleotidi, mentre le più grandi possono arrivare fino ad un milione.
Il processo di sequenziamento richiede diverse reazioni enzimatiche per arrivare a compimento. Tuttavia, lo stadio iniziale prevede sempre l’utilizzo di endonucleasi di restrizione, vale a dire degli enzimi che “tagliano” la molecola di DNA in presenza di sequenze note di quattro basi. In questo modo si ottengono filamenti di lunghezza compresa tra 150 e 100 nucleotidi. A questo punto i filamenti vengono ulteriormente degradati attraverso altri enzimi, i quali tagliano la molecola in presenza di una base precisa. Ripetendo l’operazione per ciascuna delle quattro basi su di uno stesso filamento, si ottengono filamenti di alcune unità nucleotidiche. I nuovi filamenti vengono quindi sottoposti ad elettroforesi su gel. Tramite l’elettroforesi è possibile comprendere in che sequenza si trovavano i nucleotidi quando erano formavano il DNA.
Nell’ultimo mezzo secolo sono stati fatti passi da gigante nel campo del sequenziamento del DNA: nel 1978, infatti era possibile sequenziare solo fino a 200 nucleotidi, mentre nel 1985 oltre 170.000 unità nucleotidiche. Al giorno d’oggi è possibile sequenziare diverse migliaia di unità nucleotidiche al giorno.
Fonte
- Chimica Organica, H. Hart, C.M. Hadad, L.E. Craine, D.J. Hart